class: center, middle, transition, container-fluid count: false .row[ .col-md-5.text-right[  ] .col-md-7.text-left[ # Alien ## Prédire les interactions entre espèces avec R .block[ Par Steve Vissault .small[(M.Sc)], et Kévin Cazelles .small[(Ph.D)] .small[ [Chaire de recherche en Écologie Intégrative](https://ielab.recherche.usherbrooke.ca)] ] .block[   ] ] ] --- class: slides # Mise en contexte ## Les réseaux: une façon d'abstraire le monde .row[ .col-md-4.text-center[ #### Réseaux de transports <hr style="background-color:#034370;height: 1px; border: 0;width: 75%">  <!-- TODO: mettre réseau de bus de Québec --> ] .col-md-4.text-center[ #### Réseaux sociaux <hr style="background-color:#034370;height: 1px; border: 0;width: 75%">  ] .col-md-4.text-center[ #### Réseaux écologiques <hr style="background-color:#034370;height: 1px; border: 0;width: 75%">  ] ] --- class: slides # Définition d'un réseau ## Une sémantique conjointe .row[ .col-md-6.text-center[ 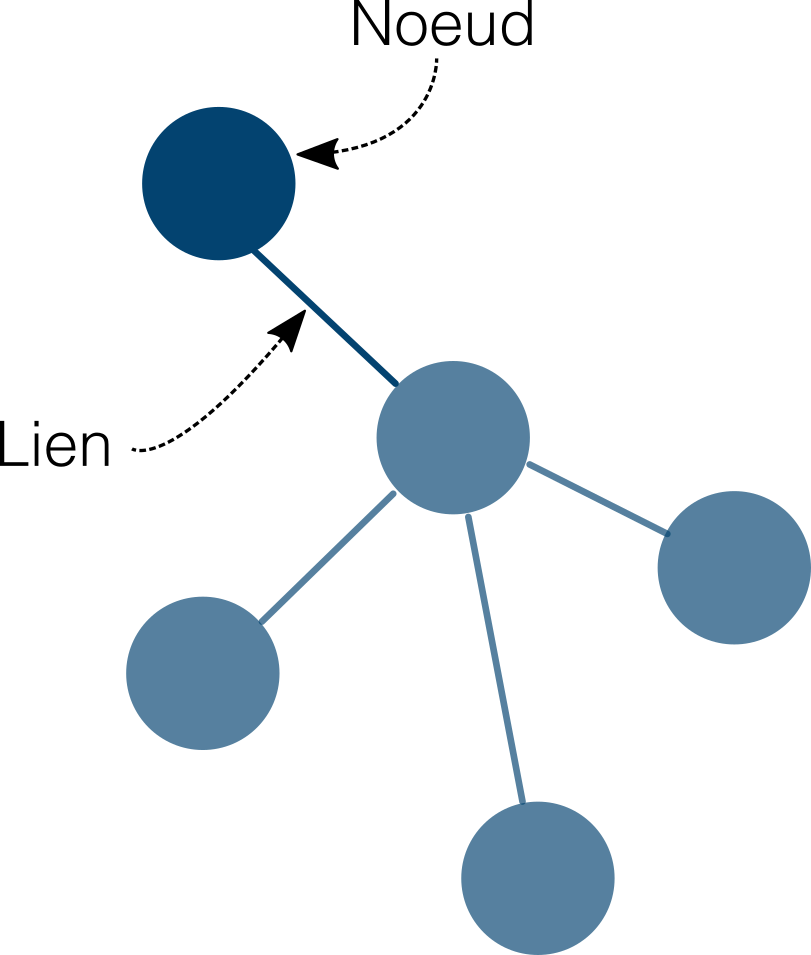 ] .col-md-6[ - Théorie des graphs - Connus dans les disciplines de l'informatique et des mathématiques - Un graph est un assemblage de **noeuds** reliés par des **liens** ] ] --- class: slides, middle, transition # Prédire à partir des propriétés --- class: slides # Les propriétés des noeuds ## Les réseaux sociaux .row[ .col-md-6[  ] .col-md-6[ Chaque membre d'un réseau dispose de propriétés qui le définisse: - Âge - Sexe - Lieu de naissance - Profession - Études etc. ] ] --- class: slides # Les propriétés des noeuds ## Comment prédire les liens à partir des propriétés des noeuds? .row[ .col-md-6[  ] .col-md-6[ Si je diffuse une nouvelle... #### Est-ce que cette nouvelle sera visible sur le fil d'actualité de mes amis Facebook (création d'un lien)? ] ] --- class: slides # Prédire un lien ## Définir un lien à partir des propriétés d'un noeud .row[ .col-md-6.text-center[  ] .col-md-6.text-left[ #### Simple algorithme: $$\sum_{lien=e} u_e w_e d_e$$ Où: - `\(u_e\)` est le *score d'affinité* entre mon ami et moi (ayant posté la nouvelle) - `\(w_e\)` le poid attribué à ce *type de nouvelle* - `\(d_e\)` le *temps* depuis la création de la nouvelle ] ] --- class: slides # Prédire un lien .row[ .col-md-12.text-center[ ## Ce type d'algorithms de rangs sont répandus à travers de nombreuses plateformes. ] .col-md-6.text-center[  ] .col-md-6.text-center[  ] ] .row[ .col-md-6.text-center[  ] .col-md-6.text-center[  ] ] --- class: slides, transition, middle .row[ .col-md-7[ # Écologie ## Peux-t-on faire la même chose en écologie? ] .col-md-5[  ] ] --- class: slides # Les propriétés des noeuds ## Les réseaux écologiques .row[ .col-md-6[ #### Réseau plantes-pollinisateur  .small[Bartomeus *et al*, 2017] ] .col-md-6[ #### Les propriétés des organismes .panel.panel-info[ .panel-heading[  Phylogénie ] .panel-body[ Indique le degré de parenté d'un organisme avec les autres organismes du réseau (distance génétique) ] ]  .small[A. Wild & K. Schaefer] ] ] --- class: slides count: false # Les propriétés des noeuds ## Les réseaux écologiques .row[ .col-md-6[ #### Réseau plantes-pollinisateur  .small[Bartomeus *et al*, 2017] ] .col-md-6[ #### Les propriétés des organismes .panel.panel-warning[ .panel-heading[  Traits ] .panel-body[ Mesure qualitative ou quantitative prise sur un organisme (i.e. Taille des ailes, Type d'appareil bucal etc.) ] ]  .small[L. Watson & M. J. Dallwitz] ] ] --- class: slides count: false # Les propriétés des noeuds ## Les réseaux écologiques .row[ .col-md-6[ #### Réseau plantes-pollinisateur  .small[Bartomeus *et al*, 2017] ] .col-md-6[ #### Les propriétés des organismes .panel.panel-success[ .panel-heading[  Co-occurence et Co-abondance ] .panel-body[ Les deux organismes doivent être présent sur le site au moment de l'échantillonnage. ] ]  ] ] --- class: slides count: false # Les propriétés des noeuds ## Les réseaux écologiques .row[ .col-md-6[ #### Réseau plantes-pollinisateur  .small[Bartomeus *et al*, 2017] ] .col-md-6[ #### Les propriétés des organismes .panel.panel-info[ .panel-heading[  Phylogénie ] ] .panel.panel-warning[ .panel-heading[  Traits ] ] .panel.panel-success[ .panel-heading[  Co-occurence et Co-abondance ] ] .panel.panel-primary[ .panel-body[ # Problématique 1 - Une grande diversité de structure et format de données - L'information sur certaines espèces peut-être manquante ] ] ] ] --- class: slides # Écologie ## Peux-t-on prédire les liens à partir de la similarité entre espèces? .row[ .col-md-12.text-center[ 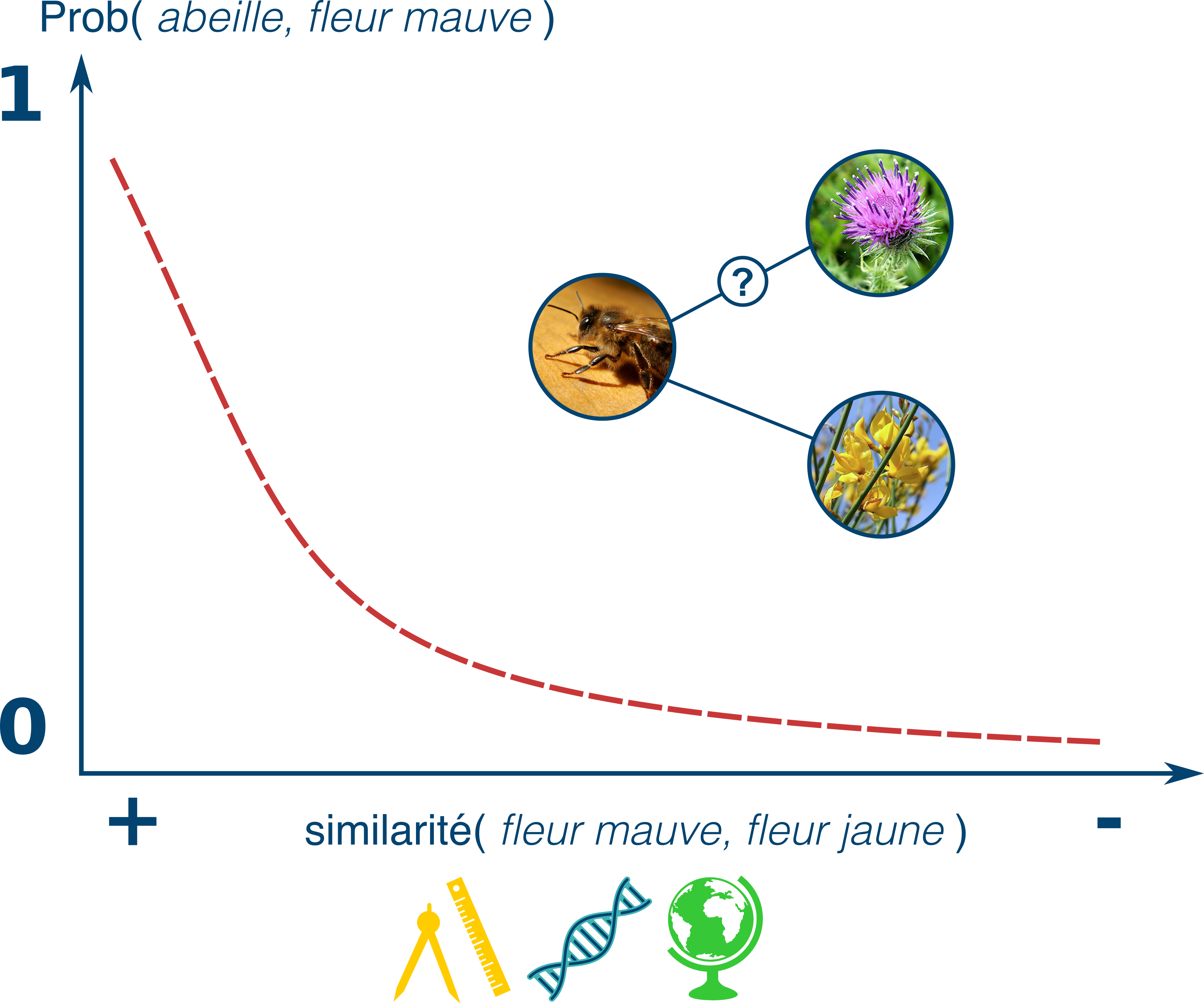 ] ] --- class: slides # Écologie ## Peux-t-on prédire les liens à partir de la similarité entre espèces? .row[ .col-md-6[ 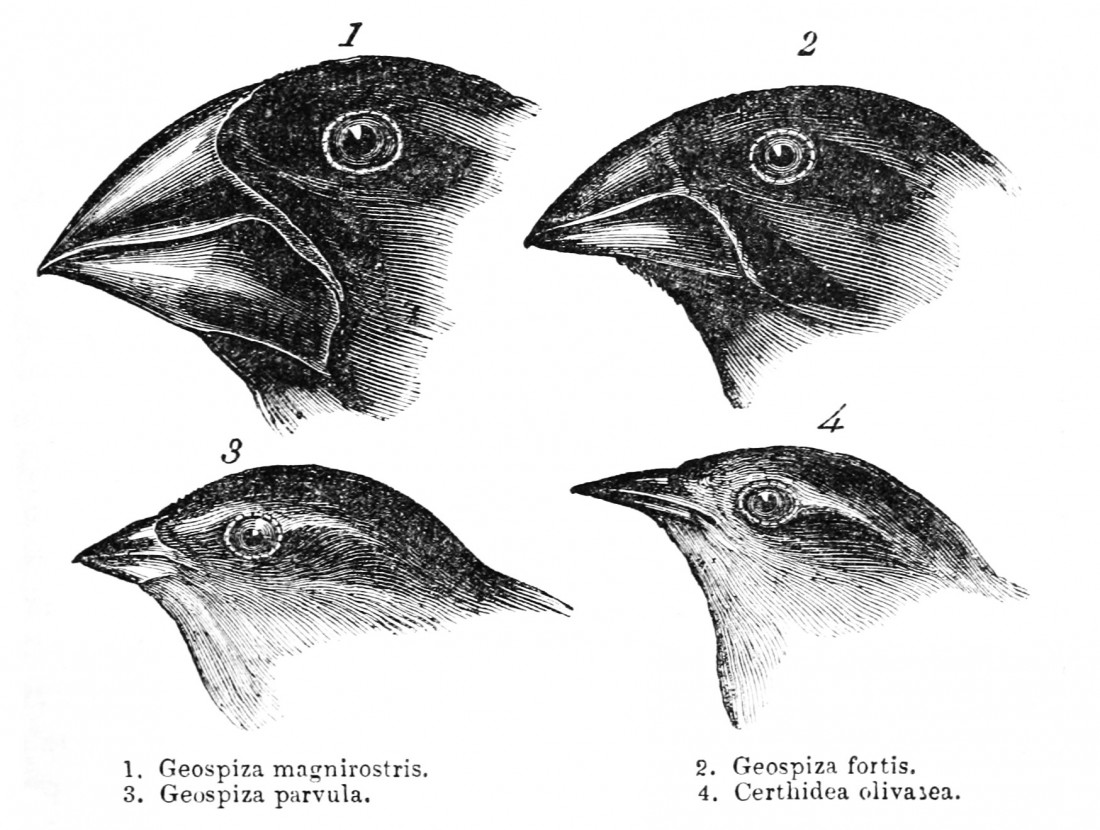 ] .col-md-6[ #### Plusieurs méthodes existent en écologie: - 2 méthodes de Machine learning *(approche informatique)* - 3 méthodes orientées par les données *(approche bayésienne)* - 6 méthodes basées sur la correspondance entre traits *(approche fréquentiste)* ] ] --- class: slides # Écologie ## Peux-t-on prédire les liens à partir de la similarité entre espèces? .row[ .col-md-6[  ] .col-md-6[ <br> .panel.panel-primary[ .panel-body[ # Problématique 2 - Pas toutes implémentées dans R - Structures de sorties divergeantes, rendant difficile la comparaison méthodologique - Intensives ] ] ] ] --- class: slides, transition, middle # Le projet Alien --- class: slides # Le projet Alien ## Pourquoi alien? .row[ .col-md-6.text-center[  ] .col-md-6[ Être en mesure d'évaluer les potentiels liens générés par l'arrivé d'une nouvelle espèce dans un réseau (*Invasive species*). - Pertinent dans un contexte de changements climatiques où les espèces vont être amenées à se déplacer. - On parle d'un ré-assemblage des communautés par perte ou gain en espèce ] ] ??? Les réseaux partagent tous une sémantique conjointe: des noeuds et des liens Vérifier graph E --- class: slides, middle # Le projet Alien... ### Une initiative d'un réseau de collaborateurs .row[ .col-md-7[ <iframe src="./network.html" width="100%" height="500px" frameBorder="0"></iframe> ] .col-md-5[ #### Développer un package R permettant de prédire les interactions entre espèces #### **Les objectifs**: - Rendre les analyses de réseaux davantage reproductible - Permettre la comparaison méthodologique ] ] --- class: slides, middle # Le projet Alien... ### Une initiative d'un réseau de collaborateurs .row[ .col-md-7[ <iframe src="./network.html" width="100%" height="500px" frameBorder="0"></iframe> ] .col-md-5[ #### Développer un package R permettant de prédire les interactions entre espèces #### **Les défis**: 1. Générer un format standard de données propre au package 2. Implémenter les différentes méthodes avec une structure de sortie uniforme 3. Développer des outils d'évaluation/diagnostique des modèles ] ] --- class: slides, middle # Le projet Alien... ### Une initiative d'un réseau de collaborateurs .row[ .col-md-7[ <iframe src="./network.html" width="100%" height="500px" frameBorder="0"></iframe> ] .col-md-5[ #### **Les bonnes pratiques du développeur:** - Intégration d'un système de contrôle de version (Git) - Utilisation de tests unitaires pour le contrôle de qualité du code (package `testhat`) - Construction du package à chaque modifications du code (`codecov`,`appveyor`,`travis CI`) - [Dépot sur GitHub](https://github.com/TheoreticalEcosystemEcology/alien) ] ] --- class: slides # Le projet Alien... ### Le package R .col-md-12[ <br> <br> <br> 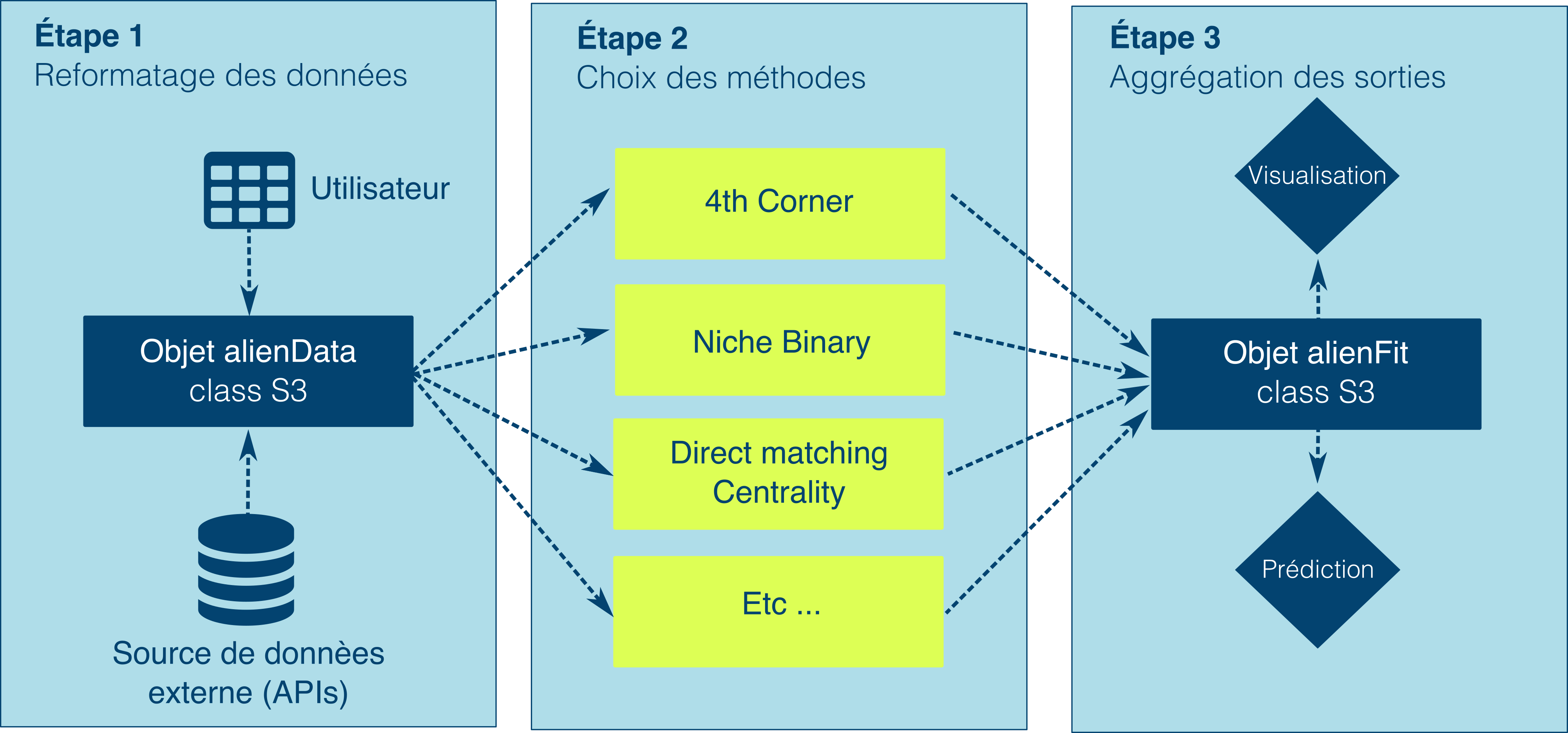 ] --- class: slides # Le projet Alien... ### Étape 1: Formatage des données #### Structure de l'objet `alienData` .row[ .col-md-6[ ```r > str(bartomeus) List of 10 $ idObs :'data.frame' $ interactPair:'data.frame' $ interactSp : int [1:331, 1:331] ... $ interactInd : int [1:2938, 1:2938] ... $ coOcc : num [1:331, 1:331] ... $ coAbund : NULL $ siteEnv : NULL $ traitSp :'data.frame' $ traitInd :'data.frame' $ phy : NULL - attr(*, "coOccSource")= chr "idObs" - attr(*, "scaleSiteEnv")= logi FALSE - attr(*, "scaleTrait")= logi FALSE - attr(*, "interceptSiteEnv")= logi FALSE - attr(*, "interceptTrait")= logi FALSE - attr(*, "class")= chr "alienData" ``` ] .col-md-6[ ### 2 jeux de données disponibles: <br> #### 1. Bartomeus *et al*, 2017 - Données collectées en Espagne (Séville) - 273 pollinisateurs - 54 plantes - 2938 individus - 1469 interactions - Phylogénie des espèces disponible ] ] --- class: slides # Le projet Alien... ### Étape 1: Formatage des données #### Structure de l'objet `alienData` .row[ .col-md-6[ ```r > str(bartomeus) List of 10 $ idObs :'data.frame' $ interactPair:'data.frame' $ interactSp : int [1:331, 1:331] ... $ interactInd : int [1:2938, 1:2938] ... $ coOcc : num [1:331, 1:331] ... $ coAbund : NULL $ siteEnv : NULL $ traitSp :'data.frame' $ traitInd :'data.frame' $ phy : NULL - attr(*, "coOccSource")= chr "idObs" - attr(*, "scaleSiteEnv")= logi FALSE - attr(*, "scaleTrait")= logi FALSE - attr(*, "interceptSiteEnv")= logi FALSE - attr(*, "interceptTrait")= logi FALSE - attr(*, "class")= chr "alienData" ``` ] .col-md-6[ ### 2 jeux de données disponibles: <br> #### 2. Kolpeke *et al*, 2017 - 30 ans de données collectées à travers l'Europe - 374 sites - 52 Saules - 91 parasites du saule - 125 Parasitoïdes ] ] --- class: slides # Le projet Alien... ### Exemples de visualisation .row[ .col-md-6.text-center[ #### GLM avec distribution de poisson (16.3%) ] .col-md-6.text-center[ #### Random Forest (0.7%) ] ] .row[ .col-md-6[ 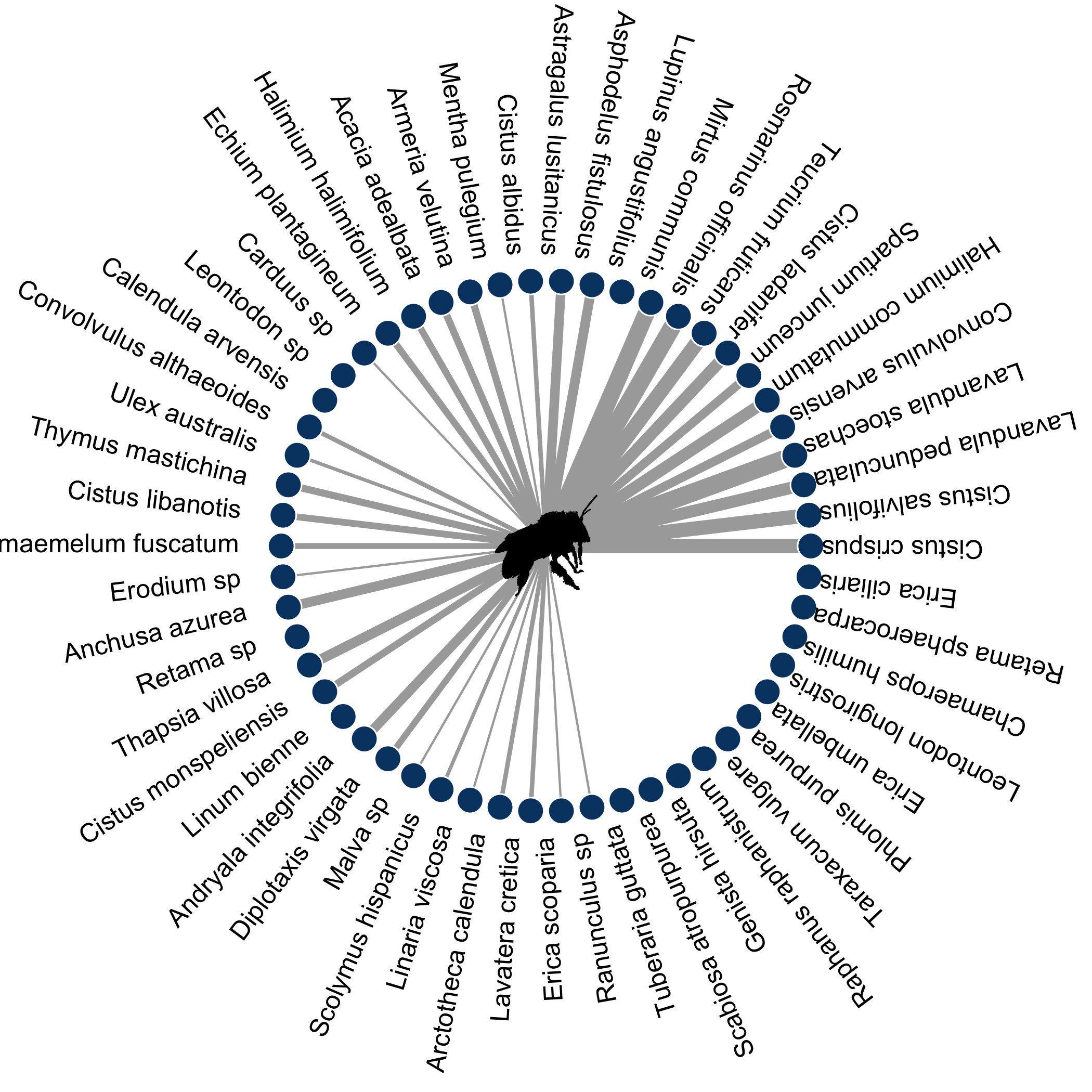 ] .col-md-6[ 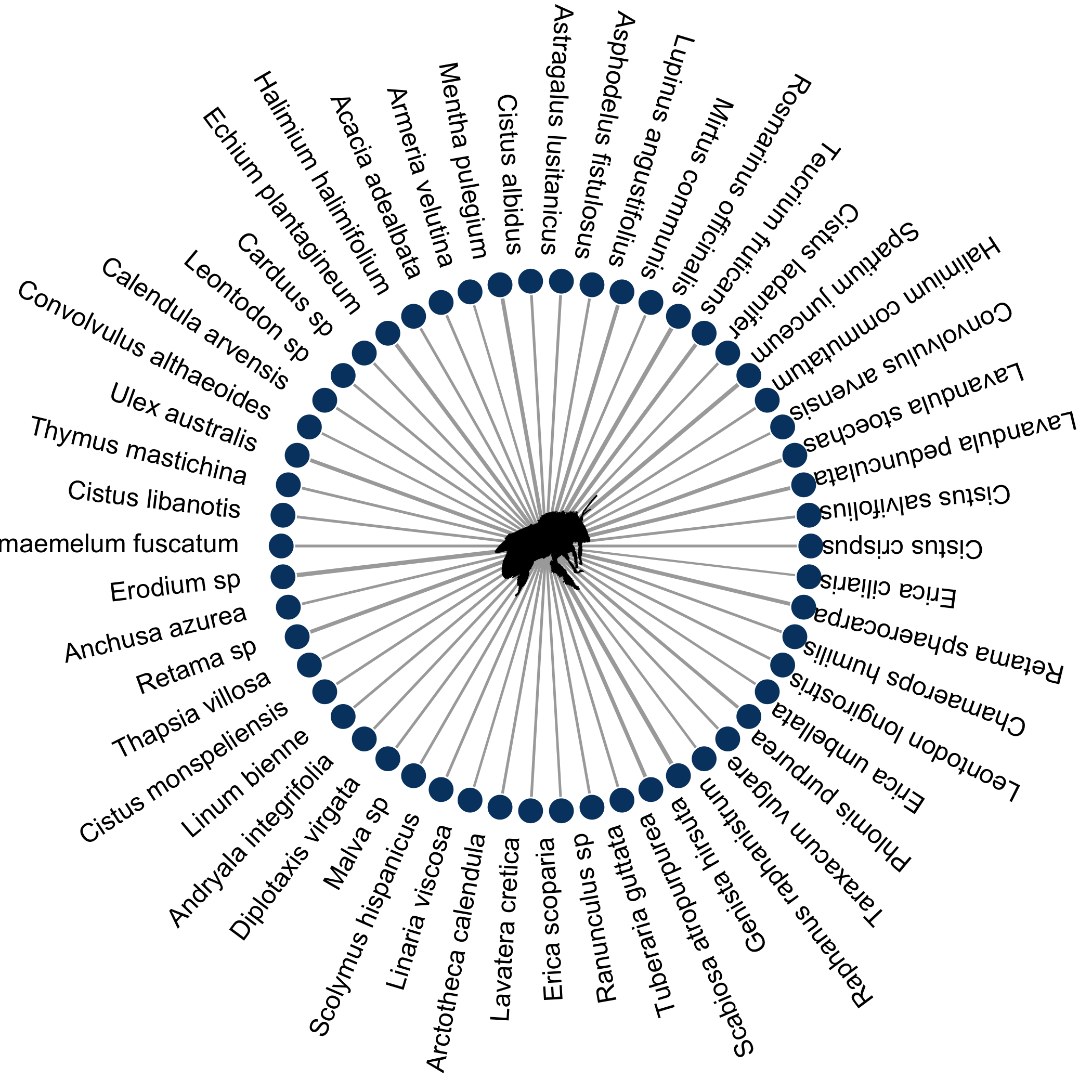 ] ] --- class: slides # Le projet Alien... ### Exemple de résultat: prédire avec Netflix .col-md-12[ <br> <br> 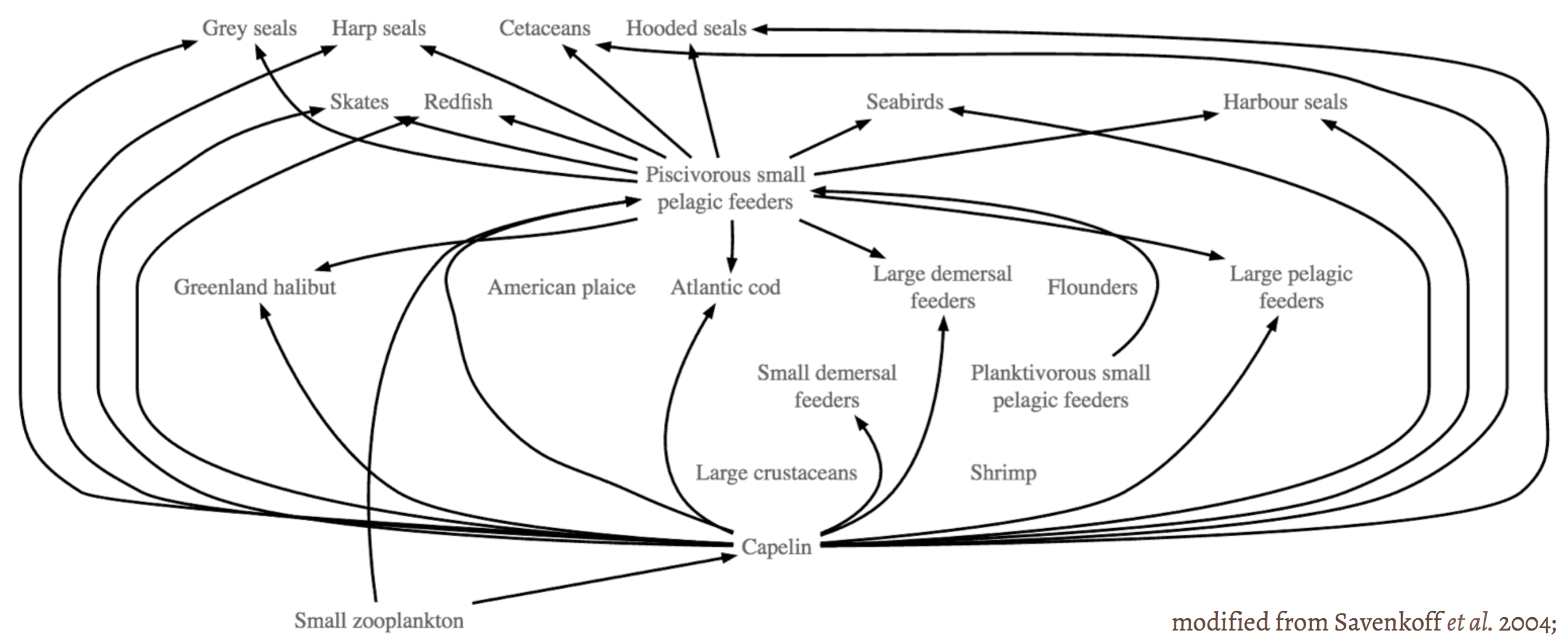 ] --- class: slides # Le projet Alien... ### Exemple de résultat: prédire avec Netflix .col-md-12[ <br> <br>  ] --- class: slides # Le projet Alien... ### Exemple de résultat: prédire avec Netflix .col-md-12[ <br> <br> 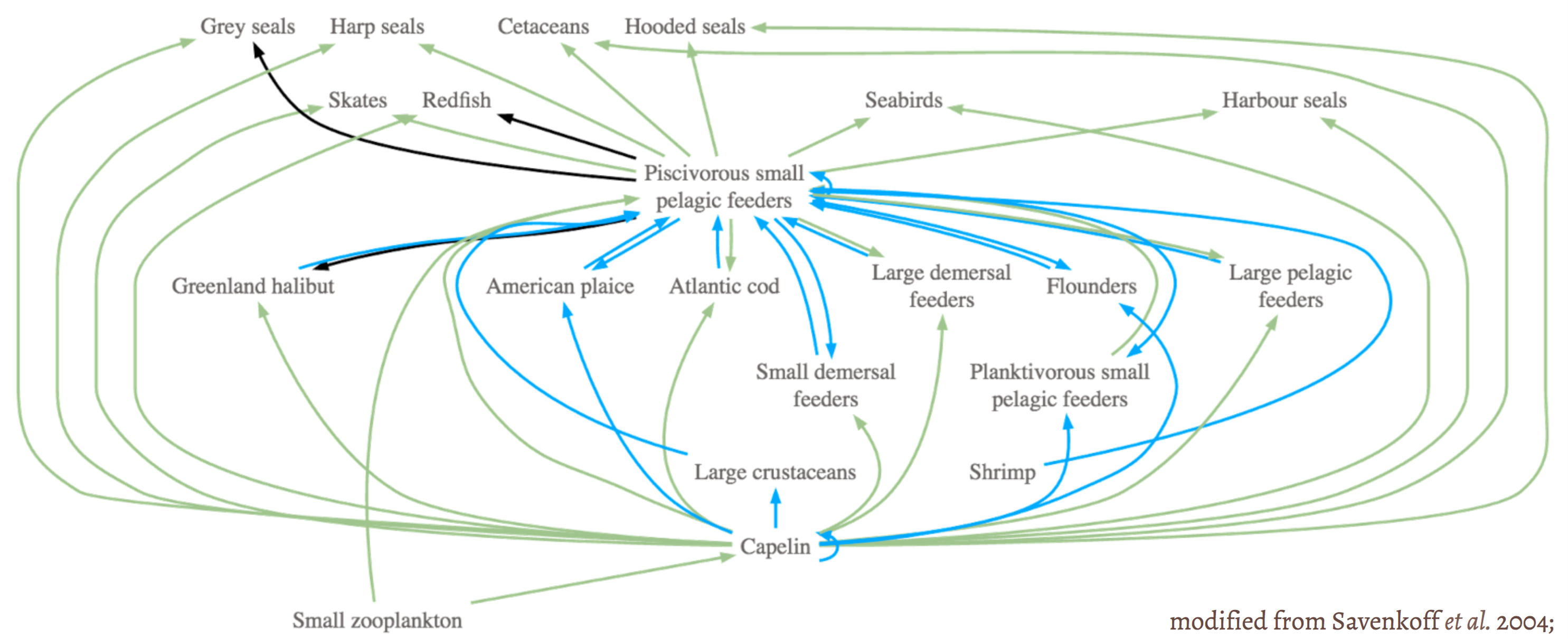  .small[ Beauchesne *et al*, 2017. ] ] --- class: slides # En résumé ## Le projet Alien... - Un effort de groupe (10 écologistes) - Package pour prédire les interactions dans un réseau écologique (11 méthodes) - Permet de reconstruire un réseau suite à l'ajout et/ou retrait d'espèces dans une communauté <br> ### Développements futurs - Décisions à venir: Objet de type S4? - Intégration des méthodes des 10 méthodes restantes - Définir la structure de sortie (objet `alienFit`) - En bref, beaucoup de travail en perspective... --- class: slides, transition, middle, center <h1 style="font-size:50px !important">Merci !</h1> --- class: slides, transition, middle, center <h1 style="font-size:50px !important">Where are we?</h1> --- class: slides # Functions implemented so far: <br/> ## Creating an `alienData` object: 1. `alienData`: to get an `alienData` object 2. `as.alienData`: conversion for certain object (data.frame, igraph) 3. `summary.alienData`: `summary` method for `alienData` objects -- <br/> ## Methods: 1. `fitDMC`: direct matching centrality 2. `fitMC`: matching centrality -- <br/> ## Outputs: 1. `alienPredicts`: Interactions predicts + Tim's paper metric + specific attributes 2. Example of proba networks metric: `getConnectance` returns the probabilistic connectance --- # Data formated so far (do we need more? *e.g.* Cohen's data): <br/> 1. `plantsPol.rda`: Batormeus data 2. `salix.rda`: Kopeleke data --- # Few comments <br/> 1. `alienData`: - `dfNodes`: species OR individuals - `dfEdges`: interactions - `dfSites`: sites - `dfOcc`: occurence => we should probably provide a function that returs the co-occurrence matrix. - `availableMethods`: given data what the user can do -- <br/> 2. Methods: - should start by checking whether the methods is in `availableMethods` - should return an `alienPredict` object -- <br/> 3. Predictions: - probability Networks - tricky questions: how to compare methods (Deviance / R-squared / McFadden) --- # What's next? <br/> ## 1. Implement other methods: 1. KNN 2. probabilistic niche model 3. What else? -- <br/> <br/> ## 2. Add more probabilistic network metrics: 1. degree 2. nestedness 3. modularity --- # A note about Harris with 3 species <br/> |State | Community | Co-occurrence | |:---------|:------------------|:----------------------| | | 1 2 3 | 1-2 1-3 2-3 | | S1 | 0 0 0 | 0 0 0 | | S2 | 0 0 1 | 0 0 0 | | S3 | 0 1 0 | 0 0 0 | | S4 | 0 1 1 **M** | 0 0 1 **N** | | S5 | 1 0 0 | 0 0 0 | | S6 | 1 0 1 | 0 1 0 | | S7 | 1 1 0 | 1 0 0 | | S8 | 1 1 1 | 1 1 1 | <br/> --  <!-- multinomial -->